Resumen
Se realizaron cuatro cultivos de linfocitos de una Hembra Ovina encontrada en las instalaciones de la Universidad Nacional de Colombia sede Bogotá, siguiendo respectivamente el protocolo de siembra y cosecha previamente establecido en el laboratorio de citogenética de la Institución. Se utilizó 1ml de sangre completa heparinizada en cada cultivo, a partir de los cuales se obtuvieron 12 láminas goteadas, asignadas de manera equitativa a los procesos de tinción Giemsa, Bandeo G (GTG) y Bandeo C (CBG). Las láminas fueron observadas en un microscopio de luz con el fin de establecer el cariotipo de la hembra ovina seleccionada, la cual cumplió con el número cromosómico normal de la especie, siendo este 2n=54. En adición a esto, no se encontraron anomalías estructurales aparentes en el cariotipo obtenido.
Introducción
La oveja domestica (Ovis aries), pertenece a la clase Mammalia, orden Artiodactyla, familia Bovidae, Genero Ovis y especie aries (Shackleton, 1997). Son animales rumiantes cuya producción va encaminada hacia las explotaciones cárnicas y textiles por su lana. Actualmente en Colombia la especie ovina presenta una baja población, contándose en total con una cifra de 1´423.274 de ejemplares distribuidos principalmente en los departamentos de La Guajira (46,69%), Boyacá (8,04%), Magdalena (7,71%), Córdoba (5,55%) y Cesar (5,41%). Estas zonas representan entonces el 73,39% de la población ovina total en Colombia (Censo Ovino y Caprino ICA 2016).
La oveja domestica cuenta con un número cromosómico diploide 2n=54 así como la mayoría de individuos en su especie (Ovis), 3 de sus pares son autosomas submetacéntricos, 23 pares son acrocéntricos y un par sexual (Ravichandran et al., 2015); sin embargo, existen algunos individuos como el Ovis ammon severtzovi (Argalí) o el Ovis nivicola (Oveja de las nieves) que cuentan con números cromosómicos diferentes: 2n=56 y 2n=52 respectivamente (Bunch, Vorontsov, Lyapunova, & Hoffmann, 1997), esto se ha atribuido a la una serie de procesos evolutivos que aún son discutidos. Lo anterior se ha sugerido en base a las evidencias citogenéticas que apuntan hacia un antecesor común entre cabras y ovejas, cuyo número cromosómico correspondería a 2n=60. A partir de ello se habla de la formación de cromosomas submetacéntricos a causa de posibles fusiones y translocaciones entre otros cromosomas (Bunch, Foote, & Spillett, 1976). Dentro de la familia Bovidae se han encontrado bandeos homólogos entre cromosomas de búfalos de agua, cabras y ovejas, e incluso cromosomas homólogos completos entre estos dos últimos. Además, en estudios realizados se han hallado segmentos homólogos en los cromosomas X y Y de las especies mencionadas, pertenecientes a dicha familia (Kozubska, Rejduch, Czech1, Babicz, & Bąk, 2012).
Una de las anomalías más comunes encontradas en las ovejas son las translocaciones Robertsonianas, detectadas principalmente entre los cromosomas 5,26; 8,11 y 7, 25 cuyas consecuencias rara vez se observan en bajas de tasas de fertilidad (Yimer & Rosnina, 2014), aun así se han detectado otras anomalías tales como translocaciones reciprocas las cuales se consideran como mutaciones de poca frecuencia en esta especie, por ejemplo, la rcp(4q;12q)(q13;q25) detectada recientemente en Italia (A. Iannuzzi et al., 2013) y la t(18;23)(q14;q26) en la cual se han encontrado consecuencias deletéreas en la fertilidad de los animales, llegando incluso estos a presentar esterilidad (Alessandra Iannuzzi et al., 2014). Por otro lado, el freemartismo es igualmente considerado como una mutación de baja aparición en los ovinos, a pesar de que la frecuencia de partos de mellizos es común, los reportes de la mutación son realmente escasos, lo que se evidencia en un estudio realizado sobre la raza Rideau Arcott en la cual se obtuvo una frecuencia del 4,35% (Brace, Peters, Menzies, King, & Nino-Soto, 2008).
Con el presente trabajo se busca entonces identificar los cromosomas y recrear el cariotipo de la especie Ovis aries, mediante la utilización de una oveja hembra ubicada en la Universidad Nacional de Colombia, sede Bogotá.
Materiales y Métodos
- Siembra y cultivo de Linfocitos T
Para la obtención del cariotipo deseado se inició con la preparación y obtención del cultivo de linfocitos T, mediante una muestra de sangre periférica equivalente a 4ml. Se seleccionó entonces una hembra Ovina de raza criolla, de 4 años y medio de edad encontrada dentro de las instalaciones de la Universidad Nacional de Colombia, sede Bogotá; para la toma de muestra se utilizaron dos tubos vacutainer a los cuales se les añadió previamente 0,05 ml de agente anticoagulante, en este caso heparina sódica. La sangre completa se obtuvo directamente de la vena yugular y tras ser extraída al vacío en los tubos se llevó al laboratorio, manteniéndola refrigerada hasta el momento de la siembra, la cual fue llevada a cabo 24h después de haber sido tomada.
Para la siembra se utilizaron 4 frascos de cultivo y 4ml de sangre heparinizada (1ml por frasco), previamente se preparó para cada frasco una solución compuesta por 8ml de medio RPMI 1640, 1ml de suero fetal bovino (SFB) y 0,5ml de fitohemaglutinina. Cada elemento (pipeta y frasco de sustrato) era flameado rápidamente antes y después de su utilización para garantizar de este modo la inocuidad de los implementos y de la solución preparada. Finalmente se homogenizó cada frasco y fueron sometidos a un periodo de incubación de 70 horas a 37°C.
Una vez cumplido dicho lapso de tiempo se procedió a cosechar. Para esto se añadieron 0,2ml de colchicina en cada frasco de cultivo, y de dejó actuar durante 1 hora, periodo en el cual los frascos vuelven a ser guardados en la incubadora.
Pasado el tiempo correspondiente los frascos fueron sacados nuevamente y vaciados en tubos de para ser centrifugados a 1200 rpm durante 10 minutos. En la primera centrifugación se eliminó el líquido sobrenadante y se re suspendió el sustrato en 8ml de solución hipotónica para llevar luego a incubación durante 30 minutos más, tras lo cual se aplicó 1ml de Carnoy. Se homogenizó el sustrato y se repitió el proceso de centrifugación y re suspensión hasta que el líquido adquirió total transparencia con posible apreciación del botón de células en color blanco en el fondo del mismo; se eliminó entonces el sobrenadante, dejando una cantidad que apenas sobrepasara el botón.
- Índice mitótico (IM) y eficiencia del mitogeno (EM)
Una vez obtenida esta cantidad se homogenizó el preparado con el cual fueron goteadas 4 láminas y posteriormente flameadas para ser luego llevadas a un periodo de secado al aire. Cabe resaltar que se guardó una parte del preparado para realizar las técnicas de bandeo cromosómico respectivas.
Para la tinción Giemsa se utilizó el respectivo colorante, aplicándolo en toda la superficie de las láminas obtenidas en las cuales se dejaron actuar durante 15 minutos luego de lo cual fueron juagadas, secadas y observadas en el microscopio utilizando el objetivo 10x para realizar el conteo de células transformadas, no transformadas y de metafases en 10 campos de las láminas seleccionados al azar para establecer el índice mitótico y la eficiencia del mitogeno de las preparaciones mediante las siguientes ecuaciones:
- Células Totales
Eficiencia del mitogeno: #Células en metafase + #Células transformadas * 100
- Células Totales
Se espera que el índice sea mayor o igual al 50% y la eficiencia presente un valor mayor o igual al 10% para ser considerados los resultados como adecuados (Barragán K., Jiménez L. y Sánchez C., 2005). A su vez se observaron las metafases encontradas en el objetivo 100x y se realizó el conteo de los cromosomas para establecer el número cromosómico de la hembra Ovina estudiada.
- Bandeo cromosómico
Bandas G (GTG): Se realizó con 4 láminas previamente goteadas a las cuales se les adicionó una disolución de tripsina en Hanks sin Ca, Mg ni rojo fenol hasta cubrir la superficie de la lámina. Se dejó actuar durante 3 minutos y posteriormente se dejó actuar 1 minutos la disolución de tripsina en Hanks con Ca, Mg y rojo fenol. Una vez hecho esto se añadió el colorante Giemsa para que actuara durante 30 minutos.
Las láminas se observaron con el objetivo 10x y fueron marcadas las metafases más claras encontradas, a partir de las cuales se seleccionó una para la elaboración del cariotipo a partir del modelo ya estandarizado (Ver Anexo 1).
Bandas C (CBG): Se sumergieron 4 láminas previamente goteadas en una solución de ácido clorhídrico durante 60 minutos, posteriormente se pasaron a hidróxido de Bario a 56°C durante 3 minutos. Luego de lavar las láminas se ubicaron en una solución salina (sodio y citrato) a 53°C durante una hora más. Finalmente se lavaron con agua destilada y se procedió a agregar el colorante Giemsa el cual actuó durante 30 minutos.
Una vez teñidas las láminas se observan bajo el objetivo 10x y se seleccionó nuevamente una de las metafases para la elaboración de un cariotipo complementario.
Resultados
La primera lámina analizada (Lámina A) para la determinación del IM y EM se descartó por el bajo número de células encontradas, las cuales no fueron superiores a 2 en los campos observados. Aun así, en comparación a otros trabajos realizados en el laboratorio, el número de células encontrados en las láminas restantes fue significativamente bajo, lo cual se le atribuye al lapso de tiempo entre toma de muestra y siembra del cultivo de linfocitos.
El número de células encontrado en los 10 campos de las 3 láminas restantes fueron:
- Lamina D:
Campo 1. CT2= 19 NT3=7 M4=0 Campo 2. CT= 21 NT= 15 M= 0
Campo 3. CT= 5 NT= 2 M=1 Campo 4. CT= 7 NT= 10 M= 1
Campo 5. CT= 9 NT= 31 M=0 Campo 6. CT= 16 NT= 24 M= 0
Campo 7. CT= 25 NT= 15 M=0 Campo 8. CT= 6 NT= 13 M= 2
Campo 9. CT= 9 NT= 13 M=0 Campo 10. CT= 9 NT= 19 M= 1
- Lamina C:
Campo 1. CT= 8 NT= 4 M= 1 Campo 2. CT= 39 NT= 21 M= 0
Campo 3. CT= 7 NT= 4 M=1 Campo 4. CT= 5 NT= 4 M= 2
Campo 5. CT= 12 NT= 9 M=0 Campo 6. CT= 15 NT= 3 M= 1
Campo 7. CT= 8 NT= 23 M=0 Campo 8. CT= 6 NT= 4 M= 1
Campo 9. CT= 13 NT= 19 M=0 Campo 10. CT= 6 NT= 6 M= 1
- Lamina B:
Campo 1. CT= 8 NT= 3 M=1 Campo 2. CT= 14 NT= 8 M= 0
Campo 3. CT= 7 NT= 6 M=0 Campo 4. CT= 11 NT= 4 M= 0
Campo 5. CT= 10 NT= 7 M=1 Campo 6. CT= 6 NT= 9 M= 0
Campo 7. CT= 5 NT= 8 M=0 Campo 8. CT= 1 NT= 10 M= 1
Campo 12 CT= 7 NT= 13 M=0 Campo 10. CT= 4 NT= 6 M= 1
Siendo así, los valores de IM y EF calculados mediante sus respectivas formulas a partir de los resultados anteriores obtenidos fueron:
| Lamina | EM (%) | IM (%) |
| B | 48,1 | 2,25 |
| C | 55,6 | 3,13 |
| D | 49,4 | 1,11 |
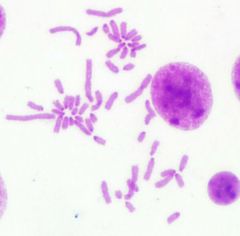
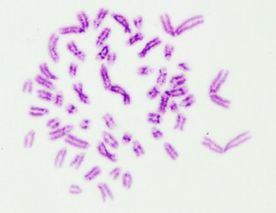
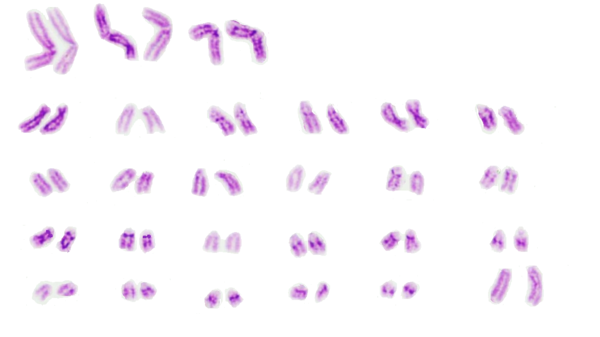
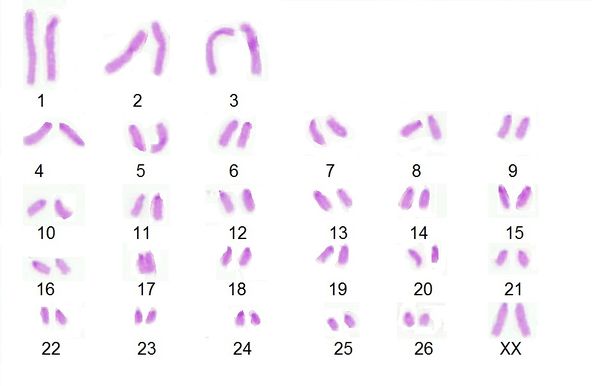
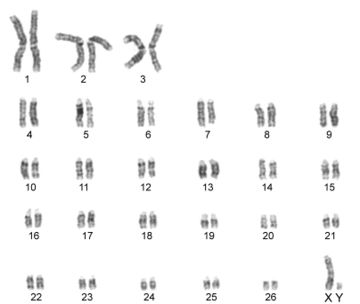
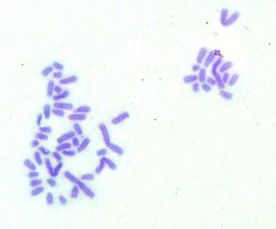
Por otra parte, las metafases obtenidas y seleccionadas de las láminas con bandas G y C que se muestran a continuación (Fig. 1 y 2) fueron ordenadas respectivamente para la obtención del cariotipo (Fig. 3 y 4):
Fig1. Metafase obtenida mediante bandeo GTG Fig2. Metafase obtenida mediante bandeo CBG
Finalmente el número de cromosomas en las metafases encontradas en dichas láminas correspondió a 54 en cerca del 90% de ellas (Ver Anexo 2 y 3).
Discusión
La EM obtenida a partir del conteo de células fue mayor al recomendado por varios autores (Barragán K., Jiménez L. y Sánchez C., 2005), aun así el IM contó con valores mucho menores a los esperados, lo cual, junto al bajo número de células encontradas por campo se le atribuye al tiempo de espera (24 horas) transcurrido entre la toma de muestra y el procedimiento de siembra de la misma.
Por otro lado, en el análisis de las metafases encontradas no se evidencian anomalías numéricas de ningún tipo, siendo esta una hembra diploide 2n=54, número cromosómico encontrado en gran parte de las metafases completas y siendo éste acorde con lo establecido por Shiwago (1931), Hansen (1973) y Ravichandran (2015) como el número cromosomico normal de la oveja domestica. En cuanto a anomalias estructurales, no se encuentran aparentemente presentes en la oveja estudiada, esto se observa en los cariotipos obtenidos con bandas G y C en los cuales se encuentra homogeneidad entre los pares de cromosomas correspondientes, ademas, la hembra no presentaba fenotipo anormal ni problemas reprodcutivos por los cuales sospechar de la presencia de alguna anomalia. Aún así las tinciones obtenidas en estos procesos no son del todo claras, puesto que las bandas obtenidas no se encuentran demarcadas en todos los cromosomas.
Conclusiones
La hembra ovina estudiada presenta un número cromosómico normal acorde a su especie siendo este 2n=54, sin presencia de anomalías numéricas ni estructurales aparentes en su cariotipo. Esto se determinó gracias a los avances que la citogenética ha tenido hasta hoy, y a partir de la cual es posible obtener herramientas prácticas para la labor de producción animal, logrando a partir de la misma detectar problemas que puedan afectar el desempeño de una producción y por tanto la economía del productor, además de brindar importantes aportes para demás disciplinas que comprometan la evolución y desarrollo de organismos vivos.
Referencias
- 1. Arslan A., Zima J. (2011) Banded karyotype of the Konya wild sheep (Ovis orientalis anatolica Valenciennes, 1856) from Turkey. Comparative Cytogenetics 5(2): 81-89 (01 Jul 2011) https://doi.org/10.3897/compcytogen.v5i2.1151
- 2. Barragán K., Jiménez L. & Sánchez C., (2005) Estandarización de las técnicas de cultivo de linfocitos y bandeo cromosómico en venado cola blanca (odocoileus virginianus). Revista de la Facultad de Medicina Veterinaria y de Zootecnia, vol. 52, núm. I, -, 2005, pp. 44-55
- 3. Brace, M. D., Peters, O., Menzies, P., King, W. A., & Nino-Soto, M. I. (2008). Sex chromosome chimerism and the freemartin syndrome in Rideau Arcott sheep. Cytogenetic and Genome Research, 120(1–2), 132–139. http://doi.org/10.1159/000118752
- 4. Bunch, T. D., Foote, W. C., & Spillett, J. J. (1976). Translocations of acrocentric chromosomes and their implications in the evolution of sheep (Ovis). Cytogenetic and Genome Research, 17(3), 122–136. http://doi.org/10.1159/000130704
- 5. Bunch, T. D., Vorontsov, N. N., Lyapunova, E. A., & Hoffmann, R. S. (1997). Chromosome number of Severtzov’s sheep (Ovis ammon severtzovi): G-banded karyotype comparisons within ovis. The Journal of Heredity, 89(3), 266–9. Retrieved from http://www.ncbi.nlm.nih.gov/pubmed/9656470
- 6. Hansen K.M. (1973) The karyotype of the domestic sheep (Ovis aries) identified by quinacrine mustard staining and fluorescence microscopy. Hereditas, 75, 233-240
- 7. Iannuzzi, A., Perucatti, A., Genualdo, V., De Lorenzi, L., Di Berardino, D., Parma, P., & Iannuzzi, L. (2013). Cytogenetic Elaboration of a Novel Reciprocal Translocation in Sheep. Cytogenetic and Genome Research, 139(2), 97–101. http://doi.org/10.1159/000346193
- 8. Iannuzzi, A., Perucatti, A., Genualdo, V., Pauciullo, A., Incarnato, D., Musilova, P., Iannuzzi, C. (2014). The Utility of Chromosome Microdissection in Clinical Cytogenetics: A New Reciprocal Translocation in Sheep. Cytogenetic and Genome Research, 142(3), 174–178. http://doi.org/10.1159/000361000
- 9. Kozubska, A., Rejduch, B., Czech1, B. D., Babicz, M., & Bąk, A. (2012). Comparative sex Chromosome hybridizations in. Ann. Anim. Sci, 12(10), 495–500. http://doi.org/10.2478/v10220-012-0041-3
- 10. Ravichandran, M. ., Saminathan, M., Aurun Prince Milton, A., Dhama, K., Suresh, C., Jeeva, K., & Misra, S. K. (2015). Comparative Cytogenetic Study of Garole and Bonpala Breeds of Sheep. Asian Journal and Veterinary Advances, 10(2), 48–61.
- 11. Shackleton, D. M. (1997). Wild Sheep and Goats and their Relatives: Status Suvery and Conservation Action plan for Caprine. (S. D. M., Ed.).
- 12. Yimer, N., & Rosnina, Y. (2014). Chromosomal Anomalies and Infertility in Farm Animals: A Review. Agric. Sci, 37(1), 1–18. Retrieved from http://www.pertanika.upm.edu.my/
Anexos:
Document information
Published on 27/07/22
Submitted on 19/07/22
Licence: CC BY-NC-SA license
Share this document
Keywords
claim authorship
Are you one of the authors of this document?