Metodología de reproducibilidad para estudios estadísticos de una población
Perla E. Castillo Flores, José Luis Fraga Almanza, Rina B. Ojeda Castañeda
Facultad de Ciencias Físico Matemáticas, UAdeC
Centro de Investigación en Matemáticas Aplicadas, UAdeC
Autor por correspondencia: perlacastilloflores@uadec.edu.mx
Resumen
En este artículo se presenta una metodología basada en la Ciencia de Datos que proporciona herramientas estadísticas y computacionales para llevar a cabo una estructuración óptima, limpieza y reproducibilidad de datos provenientes de encuestas u otros instrumentos de medición, con el fin de realizar análisis exploratorios confiables para extraer la información subyacente de los datos, que permita avalar la toma de decisiones y el establecimiento de políticas públicas frente a problemáticas de interés social. El ejemplo de aplicación de esta metodología corresponde al estudio estadístico realizado para identificar las 10 principales causas de defunción en la población infantil de los estados de Coahuila y Chihuahua, México.
Palabras clave: Reproducibilidad de datos; Limpieza de datos; Gestión de datos; Obesidad infantil; Mortalidad infantil.
2 Introducción
En las diferentes dependencias del gobierno de México, como, por ejemplo, la Secretaría de Salud, cada día se está generando un gran número de datos relacionados con el estado de salud de una población y los servicios de salud a los que tiene acceso. Esto debido a la necesidad de contar con información que les permita realizar sus actividades, tomar decisiones y establecer políticas públicas de manera eficaz y eficiente, sin embargo, para que esos datos les resulten útiles, deben ser procesados, analizados y presentados al público interesado de la forma más clara y confiable posible.
En general los datos masivos que se generan en cualquier institución o empresa, pública o privada, pueden ser procesados, analizados y presentados utilizando distintas metodologías y herramientas dependiendo de cuál sea su finalidad o utilidad.
En la literatura clásica de la estadística, la mayor parte de la teoría que se presenta, se centra en el modelado de datos, la predicción y la inferencia estadística, asumiendo que los datos se encuentran en el estado correcto para su análisis, por lo que, al mencionar las etapas del procesamiento de los datos, éstas se reducen a la obtención y clasificación de los datos de entrada, el proceso de operaciones necesarias para convertir los datos en información significativa, la presentación y diseminación de los resultados. Sin embargo, como mencionan de Jonge y Van der Loo (2013) en [1], es muy raro que los datos sin procesar, con los que se trabaja estén en el formato correcto, no tengan errores, estén completos y tengan todas las etiquetas y códigos correctos para realizar el modelado de datos, la predicción y la inferencia estadística. Por tal motivo, los usuarios y analistas de datos deben tener cuidado, tanto con las fuentes generadoras como con la información que se encuentra en éstas, desde el momento de accesar a ellas, ya que pueden tener estructuras poco entendibles o bien contener datos basura o faltantes, que les dificultará su manejo, análisis e interpretación [2].
Para llevar a cabo una buena explotación del conocimiento subyacente de grandes datos con fines de análisis para la toma de decisiones, es necesario que los datos tengan la menor cantidad posible de errores, para reducir dificultades al momento de procesarlos y que tengan la calidad necesaria para su interpretación con un alto grado de robustez y confianza. Para lograr estos requerimientos se necesita, que una vez que se ha accesado a ellos, aplicar una metodología estadística y computacional que permita organizarlos, almacenarlos y limpiarlos, mediante un procedimiento que sea repetible y documentable. Esta metodología deberá regir los procesos de acceso, depuración, complementación, y análisis de los datos que se encuentren en casi cualquier base de datos de forma confiable.
En este artículo se expone la propuesta de una metodología de reproducibilidad desarrollada para accesar, manejar y analizar bases de datos de salud de acceso libre, proporcionadas por la Dirección General de Información en Salud (DGIS) [3] del gobierno de México, con el fin de presentar resultados estadísticos de las principales causas de mortalidad de la población infantil del estado de Coahuila, así como del estado de Chihuahua, con fines comparativos. La metodología que se presenta se basa en el uso adecuado de tres herramientas computacionales: BASH, para crear una jerarquía de directorios adecuada, descomprimir archivos de forma automatizada y hacer una limpieza de archivos basura, MySQL como gestionador de bases de datos y el lenguaje R para el procesamiento de datos, comunicación con las bases de datos y el análisis estadístico de éstos. En general la metodología está basada en los principios de la Ciencia de Datos y se ofrece como una guía para realizar proyectos de análisis de datos en grandes cantidades, con el fin de que su desarrollo se vuelva óptimo, confiable y dinámico.
3 Descripción del problema
Un problema grave que surge cuando se llevan a cabo análisis exploratorios de datos, sean estos descriptivos, inferenciales o bien con fines de modelación del fenómeno o proceso a estudiar, es partir de datos sucios, incompletos, no robustos y poco confiables. Al no contarse con una metodología que permita establecer las reglas y mecanismos para realizar los diferentes procesos de acceso, estructuración y organización de los datos, así como la depuración de los mismos, los procesos posteriores para obtener los análisis estadísticos deseados, resultarán erróneos o poco robustos o no confiables. La materia prima de todo análisis exploratorio son los datos que provengan de bases bien estructuradas y limpias de errores para que se garantice que cualquier usuario interesado en reproducir los análisis o ampliarlos puedan hacerlo sin ningún problema.
Ante esta problemática se propone una metodología estadística y computacional que permite organizar, almacenar y limpiar los datos requeridos en un procedimiento estadístico y que además produzca su repetitividad y su documentación con el fin de ser transferida como herramienta para acceder, analizar y depurar casi cualquier base de datos de forma confiable.
4 Metodología
Dado un conjunto de datos, es muy importante prepararlo y limpiarlo antes de realizar cualquier análisis, de lo contrario se dispondrá de datos basura que podrían causar muchos problemas potenciales sobre todo cuando se trabaja con gran cantidad de datos [2]. Si bien es un camino largo y laborioso preparar las bases de datos, la aplicación de una metodología desde el inicio del proceso, proporcionará a los investigadores grandes beneficios, ya que permitirá automatizar las actividades de búsqueda y descarga de los datos, así como la identificación de los tipos de formatos utilizados dependiendo de la fuente, su limpieza y manipulación, para en las siguientes subsecciones hacer análisis o modelación y obtener conclusiones. En la figura 1, se presentan de forma gráfica las fases de la metodología propuesta, misma que se desarrolló e implantó en la plataforma de un sistema operativo UNIX y es útil para bases de datos estructuradas.

|
| Figura 1: Metodología aplicada, la cual se utilizó para organizar, almacenar y limpiar las bases de datos. Fuente: Elaboración propia. |
4.1 Definición de la población de estudio y búsqueda de las fuentes de información
El primer paso de la metodología consiste en definir el objetivo del estudio exploratorio y la población a ser analizada, para posteriormente buscar las fuentes de información que proporcionen los datos requeridos para lograr el objetivo de estudio en la población muestral. Se requiere hacer una búsqueda exhaustiva para encontrar las mejores y más confiables fuentes de datos para el objetivo de estudio. Este primer paso pareciera trivial, pero no lo es, es más importante conocer la confiabilidad de la fuente que el hecho de tener muchos datos.
4.2 Procesamiento de Archivos Fuentes
En la figura 2 se muestran las cuatro tareas de los scripts en Bash [4] para llevar a cabo el procesamiento de archivos. La tarea 1 permitirá tener un espacio de trabajo para guardar contenido de forma ordenada con el fin de que el acceso a los datos, figuras y demás documentos sea más sencillo, crear la jerarquía se vuelve una tarea rápida pues se puede lograr con una sóla línea de comando. La tarea 2, con wget [5] permite descargar archivos de la Web con tan sólo disponer de su URL y al mismo tiempo renombrarlos y colocarlos en los directorios correspondientes. Cuando se descargan los archivos fuente generalmente se encuentra como archivos ZIP que deben ser descomprimidos, de ello se encarga la tarea 3. Finalmente, la tarea 4 es eliminar todo el contenido que no sea útil. Para cada tarea, BASH evitará hacer trabajo de más y ahorrar tiempo volviéndose una herramienta sumamente valiosa, sin embargo, debido a que es una herramienta de los sistemas UNIX, la presente metodología no puede ser implementada en sistemas operativos de Microsoft.

|
| Figura 2: Procesamiento de archivos mediante BASH. Fuente: Elaboración propia. |
4.3 Exportación a MySQL de las Bases de Datos
Las fuentes de información elegidas proporcionan archivos que deben ser procesados para importar los datos contenidos en ellos a bases de datos con un modelo relacional y que R y MySQL permiten llevar a cabo. Primero deben crearse contenedores vacíos en MySQL que luego serán llenados con los datos contenidos en los archivos obtenidos. La creación de los contenedores se realiza mediante un script en MySQL que indica la creación de cada base de datos con el comando CREATE DATABASE [6]. Una vez que se han creado las bases de datos vacías éstas deben poblarse, la tarea puede automatizarse escribiendo un conjunto de scripts en lenguaje R. Para el entorno de trabajo se usó el software RStudio y el paquete RMySQL [7] como interfaz entre las bases de datos y R. La finalidad es mantener las bases de datos ya capturadas, limpias y en un formato específico de forma que los usuarios puedan acceder a ellas y realizar el análisis correspondiente, con ello se logrará que las bases siempre estén disponibles en el formato original para correcciones o nuevos análisis. Para establecer la conexión a la base de datos se usa la función dbConnect [8] de la librería RMySQL que permite crear funciones con los comandos e instrucciones requeridos. En los scripts de R deben definirse los paquetes a cargar, funciones y variables a ser usadas para trabajar con la información [7]. Hay diferentes funciones que permiten leer los archivos que se tienen para obtener todos sus datos dependiendo del tipo de archivo, el más común es read_csv() del paquete readr. Una vez que se ha creado la conexión con MySQL y los archivos pueden ser leídos, se pueblan las bases de datos creando las tablas correspondientes con dbWriteTable() [8]. Desde R se permite crear las bases de datos y poblarlas en las ubicaciones adecuadas según la jerarquía de directorios creada previamente. Una vez que se han importado las bases de datos a MySQL es importante ordenarlas, limpiarlas y procesarlas. Primero se debe asegurar que los datos se encuentren ordenados de forma que cada columna sea una variable y cada fila una observación, de esta forma se conseguirá un formato que consiga reducir el costo de cómputo y optimizar los resultados de los algoritmos [9]. Luego, se deben detectar los datos que son basura y desecharlos. MySQL permite realizar la limpieza de datos mediante distintas funciones que logran cambiar nombres a variables, identificar duplicados, borrar datos, etc., [10].
4.4 Análisis de Datos
En esta segunda etapa ya con las bases de datos en el formato adecuado y limpias, R permitirá transformar los datos reduciendo las observaciones a aquellas que sean de interés, ya sea creando nuevas variables que sean función de las ya existentes y/o calculando estadísticos de resumen como recuentos y medias, también permite cambiar nombres a las variables, reordenar observaciones y/o generar información mediante la visualización y el modelado de los datos. Gestionar las bases de datos mediante RMySQL permite cargarlas en una variable en R y realizar las modificaciones necesarias sin afectar a la base de datos original. Para comenzar un análisis de datos se escriben scripts en R en los que se contienen los procedimientos deseados para el análisis de los datos. Los datos específicos se obtienen mediante consultas (Querys) en MySQL por lo que será necesario crear conexión entre R y MySQL en cada script. Una forma de iniciar con el análisis exploratorio de datos es mediante la visualización, R contiene múltiples paquetes y funciones encargados de esta acción dependiendo del tipo de datos que se estén trabajando, luego pueden generarse hipótesis y probarlas.
4.5 Interfaz Shiny
Luego de que se tiene el análisis de los datos, una forma de interactuar con los resultados sin modificar el código es a través de Shiny [11], paquete de R que permite crear aplicaciones web interactivas (apps). El paquete debe ser instalado en la mayoría de los casos ya que no forma parte del core básico del lenguaje R, una forma de obtenerlo es mediante la instrucción install.package esto hará que R se comunique al CRAN (The Comprehensive R Archive Network) para obtener la librería en su versión más reciente y estable.
La figura 3 muestra la estructura de archivos al trabajar con shiny.

|
| Figura 3: Estructura de archivos shiny. Fuente: Elaboración propia. |
La figura 3 es el resultado de la creación de un proyecto en shiny por medio de RStudio (File –> NewProject –> NewDirectory –> ProyectType: Shiny Web Aplication). Es importante mencionar la naturaleza de cada uno de los archivos mostrados en la figura 3. El archivo app.R es la unión de la aplicación web y del archivo servidor encargado de procesar todos los datos involucrados en la aplicación, los directorios restantes son opcionales pero se recomienda crear los necesarios para tener un mejor orden de clasificación de los archivos. Un dato relevante es que el nombre del directorio que contiene todos y cada uno de los archivos y sub directorios de la estructura Shiny debe ser el nombre de la aplicación.
4.6 Documentación
La parte crítica de cualquier proyecto de análisis de datos es la comunicación, pues no importa que tan buenos sean los modelos y gráficos que permitan entender los datos, a menos que se puedan comunicar los resultados de su análisis a otras personas [9]. El proyecto debe ser reproducible y para ello se deben documentar los paquetes necesarios, datos y código utilizado, se recomienda cargar los paquetes al inicio de cada script en R para que sea sencillo identificarlos.
5 Un Ejemplo de Aplicación de la Metodología
Debido a que hoy en día la obesidad infantil y sus comorbilidades han cobrado relevancia en México por su alta prevalencia [12], se buscan identificar las principales causas de defunción en los infantes y si alguna o algunas de ellas pudieran estar relacionadas con el exceso de peso al ser enfermedades crónicas asociadas. Para ello se identificaron las bases de datos de defunciones que proporciona la Dirección General de Información en Salud (DGIS) [3] y se fijó como principal objetivo identificar las 10 principales causas de defunción en niños y adolescentes (menores de 16 años) del estado de Coahuila y de Chihuahua. Para obtener los resultados del análisis se siguió la metodología descrita previamente, y en esta sección se presentan algunos de los resultados obtenidos, sin embargo, no se muestra la parte de shiny ni la del documento reproducible por motivos de espacio.
5.1 Aplicación de la Metodología
Para la búsqueda de las fuentes de información y la definición de la población, se formuló la pregunta: dado el problema de obesidad infantil y las enfermedades crónicas asociadas, ¿qué datos son útiles para identificar las enfermedades por las que mayormente mueren los niños y pudieran estar asociadas a la obesidad?, luego, se definió como población a todos los menores de 16 años del estado de Coahuila y Chihuahua, ante lo que se indentificó que la DGIS [3] cuenta con un Subsistema Epidemiológico y Estadístico de Defunciones (SEED) que integra información de mortalidad del país a través de bases de datos abiertos desde el año 1998 hasta el 2018 incluyendo catálogos y descriptores de campos de cada base de datos posterior al 2012, los cuales sirven como guías que contienen los nombres de campos, tipos de datos y rango de valores. Sólo las bases posteriores al 2012 garantizan calidad al mencionar que se encuentran en la tercera forma normal, lo cual quiere decir que no hay redundancia en los datos y que las bases tiene un buen diseño en el que todos los datos son planos (strings, números y caracteres) sin estructuras multidimensionales como un arreglo o vector por lo que pueden ser leídas por cualquier gestor de base de datos.
Mediante un script en Bash se creó jerarquía de directorios que se muestra en la figura 4.
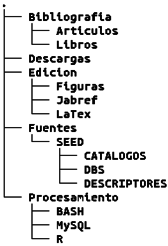
|
| Figura 4: Jerarquía de directorios. Fuente: Elaboración propia. |
La función del directorio Bibliografía fue trivial. El directorio de Descargas permitió guardar los archivos tal y como los ofrecen las fuentes. El directorio Edición se creó para guardar el documento escrito en LaTex del proyecto y para el cual se necesitaron los subdirectorios Figuras, donde se archivaron los gráficos hechos en R y Jabref para crear una base de datos de la bibliografía utilizada. Una vez que los archivos originales fueron almacenados en el directorio de descargas un archivo en Bash permitió extraer sólo lo necesario y almacenarlo según se trataba de una base de datos, un catálogo o un descriptor de campos en el subdirectorio de la fuente correspondiente, en este caso en SEED. El directorio de procesamiento almacenó los scripts necesarios para el procesamiento y análisis de datos en las herramientas Bash, R y MySQL.
Luego de crear la jerarquía de directorios, se realizó un script en Bash que permitió traer desde la fuente todas las bases de datos disponibles, guardarlas en el directorio Descargas y renombrarlas con nombres cortos (instrucciones utilizadas: wget, mv). Los archivos que se descargaron están en formato ZIP por lo que fue necesario escribir un script en BASH para descomprimir de forma recursiva los archivos obtenidos y posteriormente hacer una limpieza eliminando los archivos innecesarios dejando solo los .csv y .pdf que correspondían a las bases de datos, catálogos y descriptores de campos, mandándolos a los subdirectorios correspondientes.
Se crearon contenedores vaciós en MySQL para cada una de las bases de datos, con los nombres defun98, defun99, ..., defun17, defun18, los cuales luego fueron poblados mediante scripts en R permitiendo así tener las bases capturadas y limpias en las tablas correspondientes para que diferentes usuarios puedan disponer de ellas siempre que lo requieran a través de MySQL y R.
Con la función dbConnect de la librería RMySQL en R, se creó la conexión con MySQL para disponer de las bases de datos, la ventaja de utilizar R para llevar a cabo las consultas necesarias para el análisis es que se permite manipular a las bases de datos a conveniencia sin alterarlas directamente, por lo que no pierden su forma original con la que fueron pobladas. Una vez creada la conexión se pudieron leer los archivos requeridos a través de read.csv(). Una vez creada la conexión, se inició el análisis de los datos a través de Querys en SQL que permitieron obtener información y gráficos.
Se empezó a trabajar con bases de datos de diferentes años, sin embargo, se encontraron inconsistencias en los años 1998-2011. Los errorres encontrados fueron: falta de homogeneización en los nombres de las variables y datos inconsistentes, por ejemplo, se tenían registros de enfermedades específicas de los recién nacidos en edades posteriores al año de vida. En las bases de datos consecutivas este problema fue corregido. En la figura 5 se muestra el error antes mencionado y se observa como se distribuye el porcentaje de defunciones por la causa P220 (Síndrome de dificultad respiratoria del recién nacido) en intervalos de edad. En el año 2010, figura 5a, los resultados sugerían que hubo población que murió por dicha causa en edades posteriores al intervalo de 0-5 años de edad, lo cual es erróneo debido a las propias características de la causa P220, es decir, dicha causa no debería aparecer en edades posteriores al año de vida, tal y como sucede en el año 2015, figura 5b. Lo mismo ocurrió con otras causas del recién nacido, siendo esto un indicador de que los datos para años previos al 2012 no se encontraban bien estructurados. Además, partir del año 2012 fue posible analizar en qué tiempo murieron los infantes sin tener que redondear al año de vida, pues el dominio de la variable edad permitió ingresar minutos, horas, días y meses de vida para los menores de un año. Se decidió realizar un análisis descriptivo de las bases 2012 al 2017.
Finalmente, mediante scripts R se llevaron a cabo las consultas pertinentes a través de Querys para encontrar las 10 principales causas de defunción en los estados de Coahuila y Chihuahua en los infantes, creando gráficos y tablas de descripción de las causas, a continuación, se muestra el análisis descriptivo.
5.2 Resultados del Análisis Descriptivo
Como se indicó al final de la sección 4.1, los resultados, y la interpretación de éstos, que se presentan en esta sección están basados únicamente en el análisis de las tablas de frecuencias, graficas sectoriales e histogramas de variables relacionadas con la mortalidad infantil y las principales causas de ésta en los estados de Coahuila y Chihuahua; no corresponden a inferencias realizadas a través de métodos formales como serían el caso de las pruebas estadísticas de comparación o de métodos de asociación de variables que permiten medir la existencia de correlación entre ellas, que sería la siguiente fase en cualquier estudio estadístico de algún fenómeno poblacional.
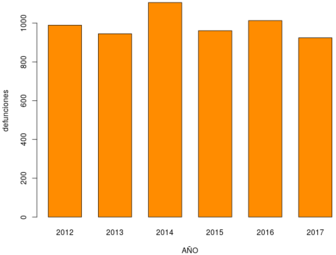
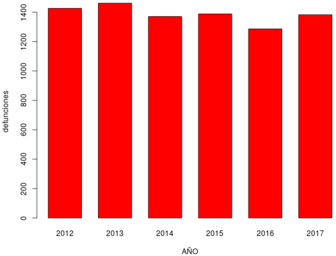
En los años analizados, figura 6, el número de defunciones se mantuvo casi constante para cada estado, en el caso de Coahuila, figura 6a, el promedio de defunciones fue de 989 y en Chihuahua el promedio fue de 1386, figura 6b. También se observa que, en Coahuila, en el año 2014, hubo un ligero incremento en el total de defunciones para el intervalo de edad analizado. Los datos para el último año del que se tiene registro (2017) indican que en Coahuila el número de defunciones bajó en comparación al año previo, sin embargo, en Chihuahua aumentó el número de defunciones en comparación al 2016.
|
|
| (a) Coahuila | (b) Chihuahua |
| Figura 6: Total de defunciones por año. Fuente: Elaboración propia. | |
Analizando el número de defunciones por intervalos de edad, se notó que no había mucha variación entre sí, pues se mantenían números muy similares y la mayoría de las causas permanecían constantes en cada año en los dos estados, por ello, aquí se presentan únicamente como ejemplo los gráficos y tablas del año 2015, por ser el año intermedio.
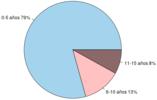
En la figura 7 se observa el número de defunciones por intervalos de edad y en la figura 8 se observa el número de defunciones por sexo, ambos en el año 2015 en el estado de Coahuila y Chihuahua. Los intervalos de edad considerados fueron los siguientes: 0-11 meses debido a las causas propias de los recién nacidos, de 1 a 5 años, de 6 a 10 años y de 10 a 15 años, figura 7. Se observa que alrededor del 70% de las defunciones corresponden a recién nacidos. Si un niño sobrevive a los primeros 11 meses de vida el riesgo a morir decrece notoriamente, destacando que el menor riesgo de perecer está en el rango de edad de 6 a 10 años. En la figura 8 se observa que en ambos estados existe ligeramente mayor prevalencia de defunciones en niños que en niñas.
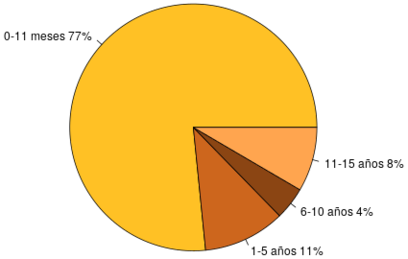
|
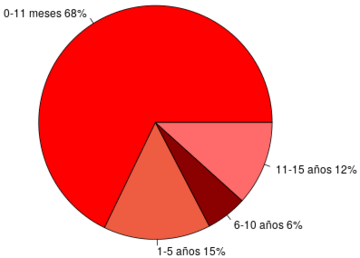
|
| (a) Coahuila | (b) Chihuahua |
| Figura 7: Total de defunciones por intervalos de edad, año 2015. Fuente: Elaboración propia. | |
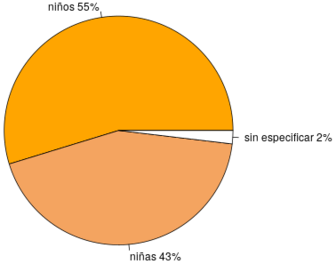
|
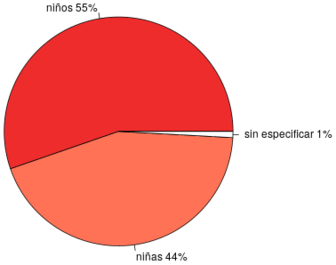
|
| (a) Coahuila | (b) Chihuahua |
| Figura 8: Total de defunciones por sexo, año 2015. Fuente: Elaboración propia. | |
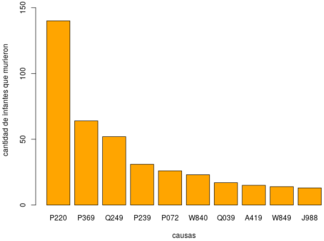
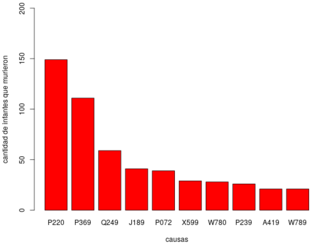
En la tabla 1 se muestra la descripción de las 10 principales causas de defunción en menores de 16 años observadas en la figura 9.
| a) Coahuila | |||
| Causa | Cantidad | Descripción | |
| 1 | P220 | 140 | Síndrome de dificultad respiratoria del recién nacido. |
| 2 | P369 | 64 | Sepsis bacteriana del recién nacido, no especificada. |
| 3 | Q249 | 52 | Malformación congénita del corazón, no especificada. |
| 4 | P239 | 31 | Neumonía congénita, organismo no especificado. |
| 5 | P072 | 26 | Inmaturidad extrema. |
| 6 | W840 | 23 | Obstrucción no especificada de la respiración en vivienda. |
| 7 | Q039 | 17 | Hidrocéfalo congénito, no especificado. |
| 8 | A419 | 15 | Sepsis, no especificada. |
| 9 | W849 | 14 | Obstrucción no especificada de la respiración en lugar no especificado. |
| 10 | J988 | 13 | Otros trastornos respiratorios especificados. |
| b) Chihuahua | |||
| Causa | Cantidad | Descripción | |
| 1 | P220 | 149 | Síndrome de dificultad respiratoria del recién nacido. |
| 2 | P369 | 111 | Sepsis bacteriana del recién nacido, no especificada. |
| 3 | Q249 | 59 | Malformación congénita del corazón, no especificada. |
| 4 | J189 | 41 | Neumonía, no especificada. |
| 5 | P072 | 39 | Inmaturidad extrema. |
| 6 | X599 | 29 | Exposición a factores no especificados que causan otras lesiones y las no especificadas. |
| 7 | W780 | 28 | Inhalación de contenidos gástricos en vivienda. |
| 8 | P239 | 26 | Neumonía congénita, organismo no especificado. |
| 9 | A419 | 21 | Sepsis, no especificada. |
| 10 | W789 | 21 | Inhalación de contenidos gástricos en lugar no especificado. |
|
|
| (a) Coahuila | (b) Chihuahua |
| Figura 9: 10 principales causas de defunción en infantes en el año 2015. Fuente: Elaboración propia. | |
Se ve en la figura 9 que las principales causas de defunción fueron: P220, P369, Q249, P072 y P239, de las cuáles, tres son propias de los recién nacidos. En el caso de Chihuahua se sumó la causa J189. La principal causa es P220 con frecuencias muy altas en comparación al resto de las causas. Como este primer análisis es marcado por la tendencia en enfermedades propias de los recién nacidos debido a que es la etapa en la que se registran la mayoría de las muertes, se llevó a cabo un segundo análisis de las causas de mortalidad en el resto de los intervalos de edad, esto se presenta en la tabla 2.
| a) 0-11 meses | |||
| Causa | Cantidad | Descripción | |
| 1 | P220 | 140 | Síndrome de dificultad respiratoria del recién nacido. |
| 2 | P369 | 64 | Sepsis bacteriana del recién nacido, no especificada. |
| 3 | Q249 | 48 | Malformación congénita del corazón, no especificada. |
| 4 | P239 | 31 | Neumonía congénita, organismo no especificado. |
| 5 | P072 | 26 | Inmaturidad extrema. |
| 6 | W840 | 19 | Obstrucción no especificada de la respiración en vivienda. |
| 7 | P240 | 13 | Aspiración neonatal de meconio. |
| 8 | Q039 | 13 | Hidrocéfalo congénito, no especificado. |
| 9 | P77X | 12 | Enterocolitis necrotizante del feto y del recién nacido. |
| 10 | J988 | 11 | Otros trastornos respiratorios especificados. |
| b) 1-5 años | |||
| Causa | Cantidad | Descripción | |
| 1 | Q039 | 4 | Hidrocéfalo congénito, no especificado. |
| 2 | V092 | 3 | Peatón lesionado en accidente de tránsito que involucra otros vehículos de motor, y los no especificados. |
| 3 | Q249 | 3 | Malformación congénita del corazón, no especificada. |
| 4 | W849 | 3 | Obstrucción no especificada de la respiración en lugar no especificado. |
| 5 | W840 | 3 | Obstrucción no especificada de la respiración en vivienda. |
| 6 | X599 | 3 | Exposición a factores no especificados que causan otras lesiones y las no especificadas. |
| 7 | A419 | 2 | Sepsis, no especificada. |
| 8 | C910 | 2 | Leucemia linfoblástica aguda [LLA]. |
| 9 | J960 | 2 | Insuficiencia respiratoria aguda. |
| 10 | C749 | 2 | Tumor maligno de la glándula suprarrenal, parte no especificada. |
| c) 6-10 años | |||
| Causa | Cantidad | Descripción | |
| 1 | G809 | 4 | Parálisis cerebral, sin otra especificación. |
| 2 | G039 | 2 | Meningitis, no especificada. |
| 3 | G049 | 2 | Encefalitis, mielitis y encefalomielitis, no especificadas. |
| 4 | V093 | 2 | Peatón lesionado en accidente de tránsito no especificado. |
| 5 | N049 | 2 | Síndrome nefrótico, no especificada. |
| 6 | V878 | 2 | Persona lesionada en otros accidentes especificados de transporte de vehículo de motor sin colisión (tránsito). |
| 7 | X094 | 2 | Exposición a humos, fuegos o llamas no especificados en calles y carreteras. |
| 8 | X950 | 2 | Agresión con disparo de otras armas de fuego, y las no especificadas en vivienda. |
| 9 | X470 | 2 | Envenenamiento accidental por, y exposición a otros gases y vapores en vivienda. |
| 10 | I490 | 1 | Fibrilación y aleteo ventricular. |
| d) 11-15 años | |||
| Causa | Cantidad | Descripción | |
| 1 | G809 | 6 | Parálisis cerebral, sin otra especificación. |
| 2 | C910 | 6 | Leucemia linfoblástica aguda [LLA]. |
| 3 | V892 | 6 | Persona lesionada en accidente de tránsito, de vehículo de motor no especificado. |
| 4 | X700 | 6 | Lesión autoinfligida intencionalmente por ahorcamiento, estrangulamiento o sofocación en vivienda. |
| 5 | E43X | 3 | Desnutrición proteicocalórica severa, no especificada. |
| 6 | C859 | 2 | Linfoma no Hodgkin, no especificado. |
| 7 | C419 | 2 | Tumor maligno del hueso y del cartílago articular, no especificado. |
| 8 | C402 | 2 | Tumor maligno de los huesos largos del miembro inferior. |
| 9 | A419 | 2 | Sepsis, no especificada. |
| 10 | G409 | 2 | Epilepsia, tipo no especificado. |
Las causas de defunción para recién nacidos observadas en la tabla 2 se mantuvieron casi constantes respecto a años previos. Las causas asociadas a lesiones o accidentes de distinta índole cobraron un alto número de vidas en los infantes de cualquier edad. En este año las causas G809 (Parálisis cerebral) y C910 (Leucemia Linfoblásticas) afectaron a infantes de los distintos intervalos de edad, aunque con mayor frecuencia cobraron la vida de aquellos en el intervalo de 11 a 15 años de edad donde fueron las dos principales causa de defunción, además G809 fue también la principal causa de defunción en el intervalo de 6 a 10 años de edad. Q039 (hidrocéfalo congénito) fue la principal causa de defunción en el intervalo de 1 a 5 años de edad, en el mismo intervalo aparecieron dos causas por obstrucción de la respiración W849 y W840. Se observó en el intervalo de los infantes mayores que la causa E43X (Desnutrición proteicocalórica severa, no especificada) apareció en la lista de las principales causas por primera vez fuera del intervalo de 1 a 5 años de edad.
Del análisis del resto de las bases de datos 2012-2017 se concluye que el Síndrome de dificultad respiratoria para el recién nacido (P220) fue la principal causa de defunción en los estados analizados, abarcando alrededor del 12% del total de defunciones en el rango de 0-5 años en Coahuila y alrededor del 9% en Chihuahua. Se detectó que las causas que cobran más vidas en recién nacidos son aquellas relacionadas con afecciones del sistema respiratorio tales como infecciones (neumonías), obstrucción respiratoria u otro tipo de trastornos repiratorios, en el caso de infantes mayores se mantienen causas relacionadas con lesiones, tumores y leucemia.
En general, el mayor número de muertes sucede dentro de los primeros 5 años de vida y disminuye para edades posteriores.
Sólo en 2016 se observó entre las causas de defunción a la diabetes mellitus, la cuál pudiera estar relacionada a la obesidad infantil, ante ello se nota que el exceso de peso no tiene afectaciones mortales en este rango de edades pues las enfermedades relacionadas pueden empezar a desarrollarse en edades tempranas pero es hasta el futuro cuando se notan las repercusiones y la mortalidad debida a estas.
6 Conclusiones
La metodología estadística y computacional desarrollada y presentada en este trabajo es de gran utilidad para todos los profesionales interesados en realizar análisis estadísticos de fenómenos demográficos, de salud, económicos y sociales, así como otra gran diversidad de procesos administrativos, a partir de bases de datos generadas con la información obtenida de encuestas u otros instrumentos de medición. Sirve como guía para llevar a cabo las requeridas etapas anteriores a la realización del análisis de exploración de los datos con el fin de garantizar la robustez y confiabilidad de éste. Las técnicas y métodos que conforman esta metodología son fundamentales para automatizar las tareas de gestión, organización, estructuración y limpieza de los datos. Además, en esta metodología se conjuntan funciones que proporciona el ambiente de software de R, para llevar a cabo análisis estadísticos descriptivos, inferenciales, de modelación, reproducibilidad y documentación [8]. Las funciones y paquetes considerados en esta metodología para la gestión y análisis de datos, son herramientas propias del sistema UNIX.
Del ejemplo de aplicación se pueden concluir dos cosas, que la estructura y depuración de una base de datos garantizan la calidad de éstos; que cada base de datos puede presentar diferentes tipos de errores y se debe estar preparado para ello, eligiendo el gestionador de bases de datos que se adecue a las características y necesidades de éstas para así seleccionar la mejor. Específicamente se deben buscar fuentes que proporcionen bases de datos bien documentadas con catálogos, descripción de su metodología, modelo y estructura que evitará invertir mucho tiempo para conocer su contenido. Las bases de datos que ofrece la DGIS tienen errores a pesar de venir de una fuente confiable. Los principales errores se encontraron en las bases previas al 2012, ya que son bases no estructuradas, y tienen inconsistencia en los nombres de las variables con respecto a las bases posteriores a esta fecha.
BIBLIOGRAFÍA
[1] De Jonge E., Van der Loo. (2013) "An introduction to data cleaning with R". Statistics Netherlands Grafimedia
[2] Ricardo Catherine. (2009) "Bases Datos". Mc Graw Hill
[3] DGIS. (2020) "Defunciones. Datos Abiertos"
[4] Ramey C. and Fox B. (2019) "Bash Reference Manual". Free Software Foundation, Inc.
[5] . (2018) "GNU Wget 1.20 Manual". Free Software Foundation, Inc.
[6] . (2020) "Section 3.3.3 Creating and Selecting a Database (MySQL 8.0 Reference Manual)". MySQL
[7] Jiménez Chura Adolfo C. (2017) "RMySQL para el análisis de datos de postulantes e ingresantes del área biomédicas a la Universidad Nacional del Altiplano (Puno Perú)", Volume 19. Revista de Investigaciones Altoandinas 2 201-210
[8] . (2019) "R Database Interface: Package DBI". CRAN R PROJECT
[9] Grolemund G. and Wickham H. (2017) "R for Data Science: Import, Tidy, Transform, Visualize, and Model Data". O Reilly, 1 st Edition
[10] DuBois Paul. (2013) "MySQL". Addison-Wesley Professional, 5th Edition
[11] RStudio Inc. (2020) "Shiny from R studio" https://shiny.rstudio.com/tutorial/
[12] Ávila et al. (2016) "Estado de nutrición en población escolar mexicana que cursa el nivel de primaria". Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
Document information
Published on 21/12/20
Submitted on 21/12/20
Licence: CC BY-NC-SA license
Share this document
Keywords
claim authorship
Are you one of the authors of this document?